논문 제목 : Deep Neural Networks for Anatomical Brain Segmentation
- Brebisson, Montana
- 2015
- 논문 링크
Summary
- SegNet은 3D 뇌 이미지를 부위별로 구별하는 segmentation 알고리즘이다.
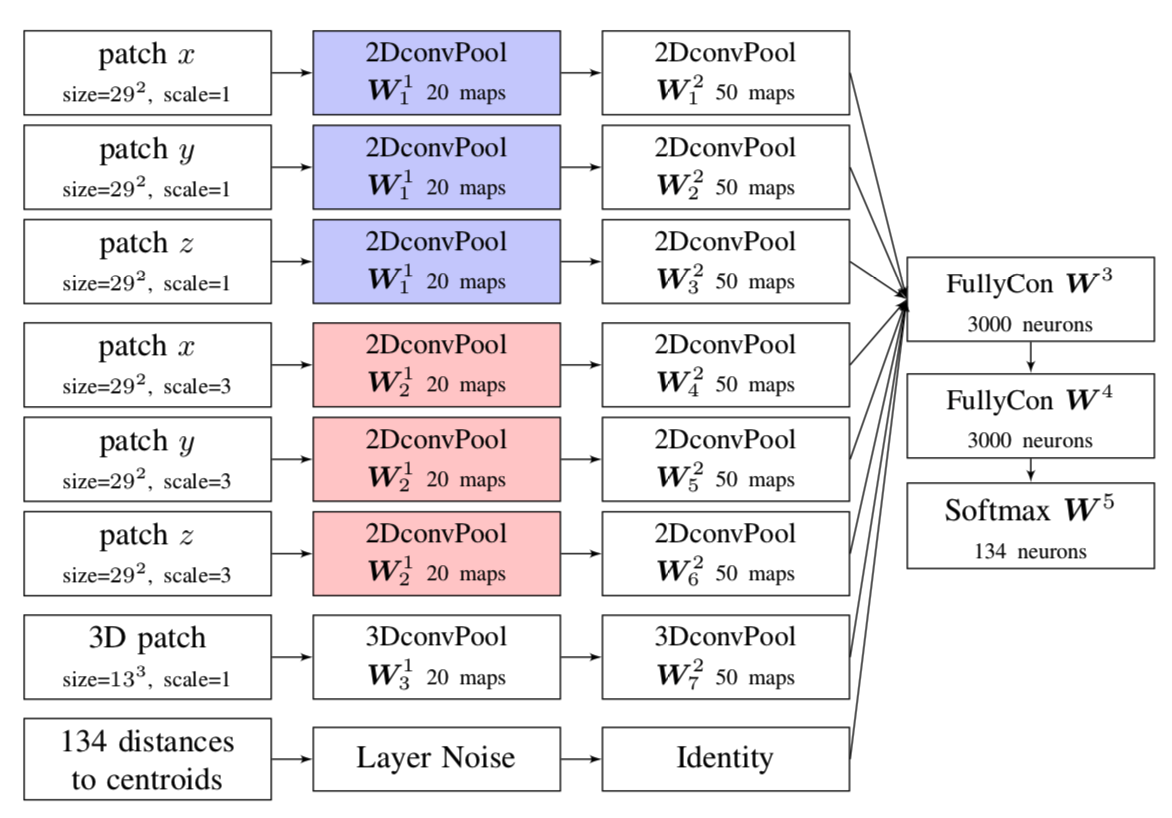
- 이를 위해 각 voxel을 중심으로 하는 3D patch와 6 종류의 2D patches, 그리고 각 부위 centroid와의 거리 값까지 총 8개의 데이터를 입력으로 사용한다.
- 이미지 patch 에서 voxel intensity 정보를, centroid와의 거리에서 상대적인 좌표 정보를 얻으므로 보다 정확한 분류가 가능해진다고 한다.
Purpose
뇌를 구성하는 각 부위의 크기는 알츠하이머 병, 파킨슨 병과 같은 뇌 질환과 연관되어 있다고 한다. 이러한 점에서 뇌 이미지를 부위별로 구분하고 그 크기를 측정하는 것은 뇌 질환을 진단하는 중요한 수단이다. 하지만 이렇게 부위별로 나누는 작업은 의사들이 직접 수행하는 경우가 많았고, 그렇기 때문이 오래 걸리고 비용이 많이 소요된다는 문제가 있다.
논문은 이러한 문제의식에서 출발하며, 이를 위해 빠르고 정확한 뇌 부분 분할을 가능하게 하는 딥러닝 기반 모델을 제시하고 있다. 구체적으로 뇌의 3D 이미지를 구성하는 각 voxel을 해부학적 부위에 따라 classification 하는 3D brain semantic segmentation 알고리즘을 제시한다.
Method
논문에서 제시하는 알고리즘 SegNet의 가장 큰 특징은 하나의 voxel에 대해 3D patch, 2D patch, 그리고 각 부위별 centroid를 입력으로 받는다는 것이다. 여기서 2D patch는 6개이기 때문에 총 8개의 입력을 받는다. 이 중 3개의 2D patch와 3D patch는 local precision을 위한 것이고, 나머지 3개의 2D patch와 centroid는 global spatial consistency를 위한 것이라고 설명한다.
Features to ensure local precision
하나의 voxel의 부위를 판단함에 있어 정교함을 높이기 위해 SegNet은 다음 네 가지 정보를 입력으로 받는다.
- 3D patch of size (a,a,a) centered on the voxel
- three 2D orthogonal patches of size (b,b) centered on the voxel
즉, 판별하고자 하는 voxel을 중심으로하는 2D, 3D patch 들이며, 이때 2D patch는 차원의 수만큼 3개를 구할 수 있어 직교하는 모든 이미지를 사용하겠다는 것이다.
3개의 2D 패치들을 사용하는 것과 관련하여 논문에서는 1개의 2D patch를 사용하는 경우와 3D patch를 사용하는 경우를 절충한 것이라고 설명한다. 즉, 공간적인 특성을 잡을 수 있으면서도 컴퓨터 자원을 적게 사용하는 방법이라는 것이다.
Features to ensure global spatial consistency
지엽적인 정교함과 더불어 넓은 영역에서 차지하는 위치 등을 고려하면 보다 정확도를 높일 수 있을 것이다. 특히 뇌 이미지의 경우 동일한 부위는 이미지별로 비슷한 위치에 있을 가능성이 높기 때문에, 다른 unstructured segmentation task와 비교해 상대적인 위치가 더욱 중요하다. 이를 위해 downscaled 2D patch와 각 부위 centroid와의 거리를 입력으로 사용한다.
- three large but downscaled 2D orthogonal patches
- 134 distances to centroid of each regions
전체적인 위치를 고려하겠다고 한다면, 가장 먼저 떠오르는 방법은 보다 넓은 영역을 보는 것이라고 할 수 있다. 하지만 단순히 patch를 입력으로 전달할 경우 메모리 공간 이슈 등 비효율성이 높다고 보고, 다운 스케일링을 진행하여 local precision을 위해 구성한 2D patch와 동일한 크기로 만드는 과정을 추가하고 있다.
즉, 본래 large patch의 크기가 (sc, sc) 였다면, s x s mean-pooling을 이용해, (c, c) 크기로 줄여 사용하겠다는 것이다.
Distance to centroid
large 2D patch에 그치지 않고 각 부위의 centroid 와의 거리 정보도 사용하겠다는 것은 voxel intensitiy 뿐만 아니라 voxel의 좌표 정보도 사용하겠다는 것으로 이해할 수 있다. 이때 절대적인 좌표값을 사용하지 않는 것은 image의 특성에 큰 영향을 받기 때문이다. 즉, 이미지 상에 뇌의 위치, 회전 정도에 따라 동일한 voxel이더라도 절대적인 위치가 크게 달라질 수 있다는 것이다.
반면 각 부위의 centroid 와의 거리를 사용한다면 이러한 문제가 없어진다. 즉 centroid 와의 거리를 상대적인 좌표로 보겠다는 것이다.
이미지 \(I\) 상 어떤 부위 \(l\)의 centroid \(c_l = (x_l, y_l, z_l)\)은 다음과 같이 구해진다.
\[c_l = { \Sigma_{v \in I^{-1}(l)}v \over \lvert I^{-1}(l) \rvert }\]Estimation of the Centroid
Train set의 경우 각 voxel의 label 정보를 알고 있으므로 부위 별로 정확한 centroid를 구할 수 있다. 하지만 label이 없는 정보에 대해서는 어떻게 centroid를 구할 수 있을지가 문제된다. 논문에서는 다음 작업을 반복하여 centroid의 근사치를 구할 수 있다고 한다.
- Centroid를 사용하는 영역 없이 네트워크에 이미지를 통과시킨다. 그 결과 voxel 별로 임시의 label을 구할 수 있고, 이를 통해 approximate centroid 도 구할 수 있다.
- approx centroid를 사용하여 다시 네트워크에 이미지를 통과시킨다. 이렇게 하면 보다 정교한(refined) label을 구할 수 있을 것이고 보다 나은 approximate centroid 또한 구할 수 있다.
이를 각 centroid가 일정 수준 수렴할 때까지 반복하는 방식으로 정확한 centroid를 구하겠다는 것이다. 이와 관련하여 논문에서는 모델이 정확하면 정확할수록 빠르게 수렴한다고 언급하고 있다.
Architecture
SegNet의 구조는 다음과 같다. 앞에서 설명한대로 8개의 입력값을 받고 있으며, 이를 제외하면 CNN 모듈과 FC 모듈로 구분되는 전형적인 구조를 띄고 있다.
Additional Study
용어 정리
- Voxel : 부피를 의미하는 Volume과 픽셀 Pixel의 합성어로, 3차원 공간에서 정규 격자 단위의 값(wiki-복셀)을 의미한다. 즉 3차원 이미지를 표현하기 위해 사용되는 픽셀을 의미한다.
- Magnetic Resonance(MR) image : 자기 공명, 자기장과 고주파를 통해 신체의 수소 원자핵을 공명시켜 나오는 신호를 측정하여 만든 이미지. MRI가 대표적이다.
- brain atlas : a brain atlas is composed of several sections along different anatomical planes of brain (wiki-brain atlas). 한마디로 뇌의 여러 단면을 모아둔 것이라고 할 수 있다.